CapTrap-seq
-
- Home
-
- 2 of 2
El genoma —la seqüència de DNA que es troba als cromosomes dins les cèl·lules— codifica les instruccions que determinen les característiques biològiques dels éssers vius. Aquestes instruccions estan específicament localitzades en regions del genoma que anomenem gens (en el cas del genoma humà es comptabilitzen uns 60.000 gens). Quan aquestes regions s’activen es transcriuen a molècules d’àcid ribonucleic (RNA), les quals, acabaran donant lloc a les proteïnes, que són els components funcionals i estructurals de les cèl·lules.
El tipus i l’estat de les cèl·lules (per exemple, en el cas de malaltia) són, doncs, determinats pel seu contingut d’RNA, és a dir, per l’abundància de les molècules d’RNA (els transcrits) que s’originen als gens (l’anomenada expressió dels gens). L’obtenció de la seqüència dels RNA que existeixen a les cèl·lules en un moment determinat (RNAseq, és a dir, la seqüenciació de l’RNA), és, en conseqüència, la millor manera de determinar l’estat cel·lular.
Fins fa poc, els mètodes d’RNAseq només permetien obtenir seqüències molt curtes, de longitud molt inferior a la dels transcrits, la qual cosa feia difícil identificar i quantificar amb precisió l’expressió dels gens. Recentment hom ha desenvolupat mètodes d’RNAseq de lectura llarga (long-read RNAseq, en anglès) que permeten, en principi, obtenir la seqüència completa dels transcrits.
Tanmateix, encara hi ha reptes importants que cal superar per tal d’obtenir aquestes seqüències, com per exemple la creació de les llibreries necessàries per a la seqüenciació de l’RNA després d’extreure’l de les cèl·lules.
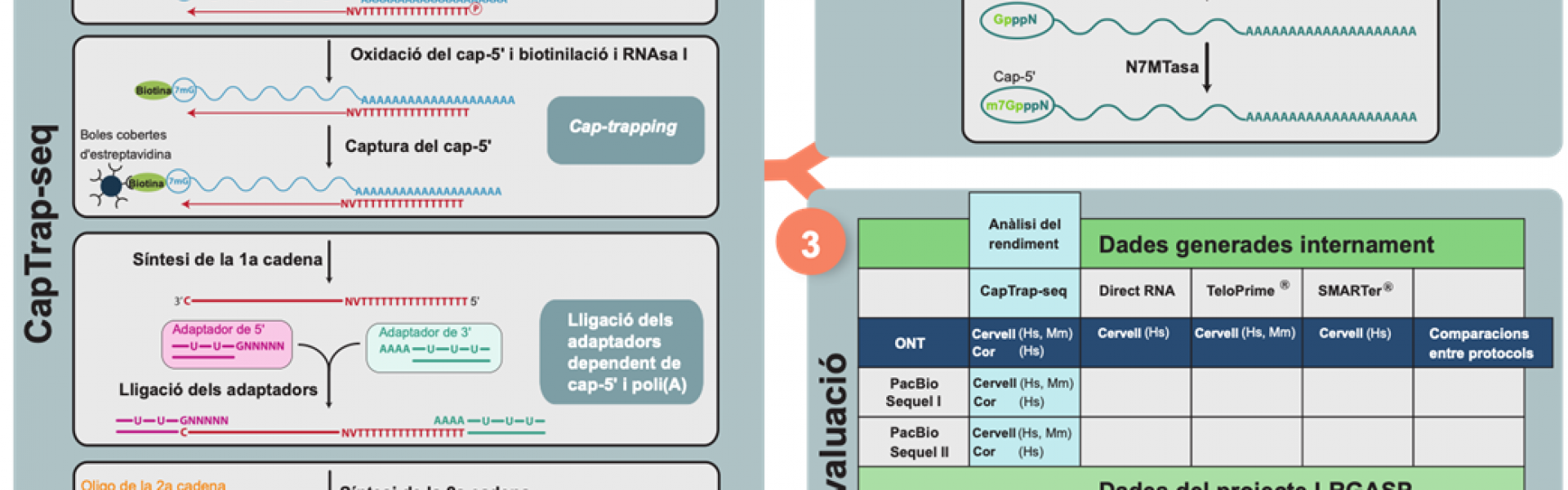
En aquest sentit, el nostre equip ha desenvolupat un mètode per crear llibreries: el CapTrap-seq (Figura 1). Aquesta tècnica millora la manera com s’extreu i es prepara l’RNA abans de ser seqüenciat. De fet, CapTrap-seq combina dues estratègies: la Cap-trapping (o atrapador de caps), per capturar els transcrits des del seu inici, i l’oligo(dT), per capturar els transcrits des del seu final.
Per validar aquesta nova tècnica, hem avaluat el funcionament de CapTrap-seq en cèl·lules obtingudes de teixits humans i de ratolí i l’hem comparat amb altres mètodes de preparació d’RNA. A més, hem utilitzat dues plataformes de seqüenciació de lectura llarga —l’ONT (Oxford Nanopore Technologies) i la PacBio (Pacific Biosciences) — per verificar la precisió de CapTrap-seq a l’hora de mesurar l’abundància dels transcrits (és a dir, l’expressió dels gens).
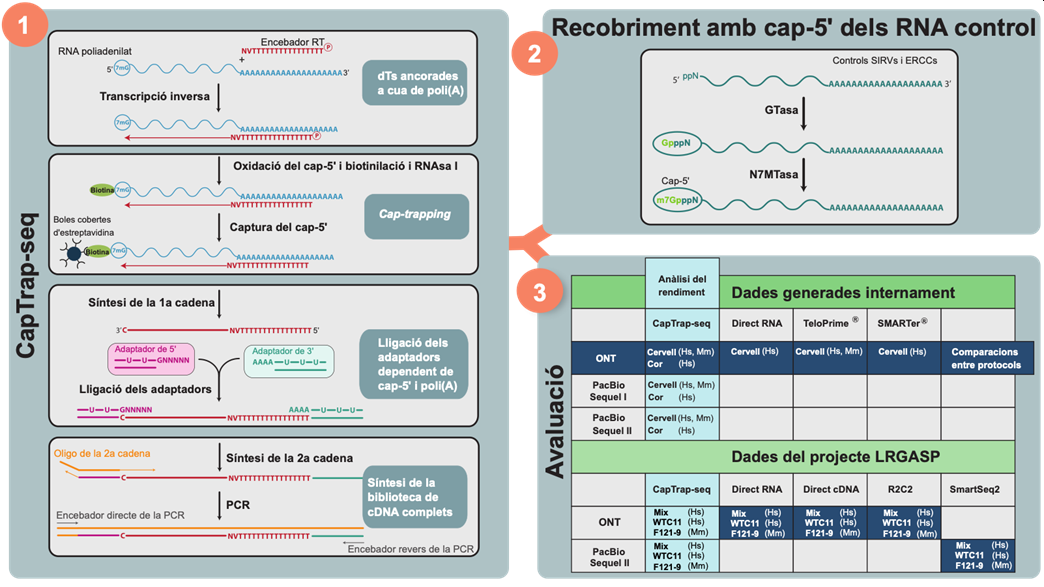
Figura 1. (1) Flux de treball del CapTrap-seq per enriquir transcrits de longitud completa des de l’extrem 5’ fins a l’extrem 3’. (2) Recobriment amb cap-5’ dels RNA control: esquema de la nova estratègia dissenyada per afegir un recobriment a les seqüències dels RNA sintètics (controls), (3) Avaluació de CapTrap-seq: protocols alternatius amb els quals hem comparat CapTrap-seq en dues plataformes diferents de seqüenciació: PacBio (Sequel I i II) i ONT, en dades produïdes a partir de teixits d’origen humà (Homo sapiens, Hs) i de ratolí (Mus musculus, Mm). Algunes d’aquestes dades han estan generades internament i d’altres han estat generades com a part del projecte LRGASP (https://www.gencodegenes.org/pages/LRGASP). Mix és una barreja de cèl·lules mare embrionàries i cèl·lules d’origen endodèrmic. WTC11 són cèl·lules mare pluripotents. F121-9 són cèl·lules mare embrionàries.
Els resultats del nostre estudi demostren que CapTrap-seq és un mètode competitiu per a la preparació de l’RNA, i que pot ser utilitzat indistintament en diferents plataformes de seqüenciació, la qual cosa el fa flexible i adaptable a les necessitats dels investigadors. Actualment ja s’està utilitzant la tècnica CapTrap-seq dins el projecte GENCODE, el consorci internacional que produeix el mapa detallat dels gens i transcrits en els genomes humà i de ratolí.
Tanmateix, cal reconèixer que tot i que CapTrap-seq ofereix molts avantatges, també té certes limitacions. En primer lloc, requereix una quantitat més gran d’RNA que altres protocols. En segon lloc, tendeix a produir seqüències més curtes. I en tercer lloc, té com a objectiu principal la identificació d’un tipus determinat de molècules d’RNA, les quals constitueixen només una fracció (biològicament molt important, això sí) de totes les molècules d’RNA que es troben a les cèl·lules.
En conclusió, la seqüenciació d’RNA de lectura llarga, juntament amb les eines d’anàlisi de dades adequades, entre les quals CapTrap-seq, té el potencial de produir per primera vegada mapes de transcrits acurats dels genomes dels éssers vius i estimacions precises de l’expressió gènica en tipus cel·lulars diferents. Això és especialment important perquè hi ha dos grans projectes de recerca internacionals en marxa en els quals els centres de recerca del nostre país tenen un paper rellevant: l’Earth BioGenome Project, per seqüenciar els genomes de totes les espècies eucariotes de la Terra, i el Biodiversity Cell Atlas, per identificar-ne tots els tipus cel·lulars).
Nota:
Aquest article és una adaptació de l’abstrat a Carbonell-Sala et al. “CapTrap-seq: A platform-agnostic and quantitative approach for high-fidelity full-length RNA sequencing”, Nature Communications (2024), https://doi.org/10.1038/s41467-024-49523-3. Autors: Silvia Carbonell-Sala1, Tamara Perteghella1,2, Julien Lagarde1,3, Hiromi Nishiyori4, Emilio Palumbo1, Carme Arnan1, Hazuki Takahashi4, Piero Carninci4,5, Barbara Uszczynska-Ratajczak1,6, Roderic Guigó1,2.
- Center for Genomic Regulation (CRG), Barcelona Institute of Science and Technology, Barcelona, Catalunya, Espanya
- Universitat Pompeu Fabra, Barcelona, Catalunya, Espanya
- Flomics Biotech, SL, Carrer de Roc Boronat 31, 08005 Barcelona, Catalunya, Espanya
- Laboratory for Transcriptome Technology, RIKEN Center for Integrative Medical Sciences (IMS), Yokohama, Kanagawa, Japó
- Human Technopole, Milà, Itàlia
- Institute of Bioorganic Chemistry, Polish Academy of Sciences, Poznan, Polònia